(图文|潘伊茹 编辑|信息 审核|张泽宇)近日,生物信息领域重要期刊Briefings in Bioinformatics接收了我校生物信息2021级本科生潘伊茹和人工智能2022级本科生吉星宇的研究论文。该论文题为Contrastive Signed Graph Diffusion Network for Predicting Crop Gene-phenotype Associations,主要开发了符号图神经网络工具CSGDN,用于基因-形状正负关联预测问题。
遗传学研究中,基因与表型之间的调节关联非常重要。全基因组关联分析(GWAS)通过鉴定基因组变异与表型的关系,揭示了数量性状位点(QTL)与表型特征之间的关联。然而,由于QTL的大小和连锁不平衡衰减距离,针对特定QTL的候选基因鉴定变得困难。转录组水平的GWAS(TWAS)能通过分析基因表达与表型变异的关联来识别关键基因,但它需要大量样本数据,导致实验成本较高。此外,TWAS方法仍依赖于关联分析框架,预测低变异频率基因的能力有限,且容易受到系统性偏差、覆盖偏差和实验批次效应等因素影响。
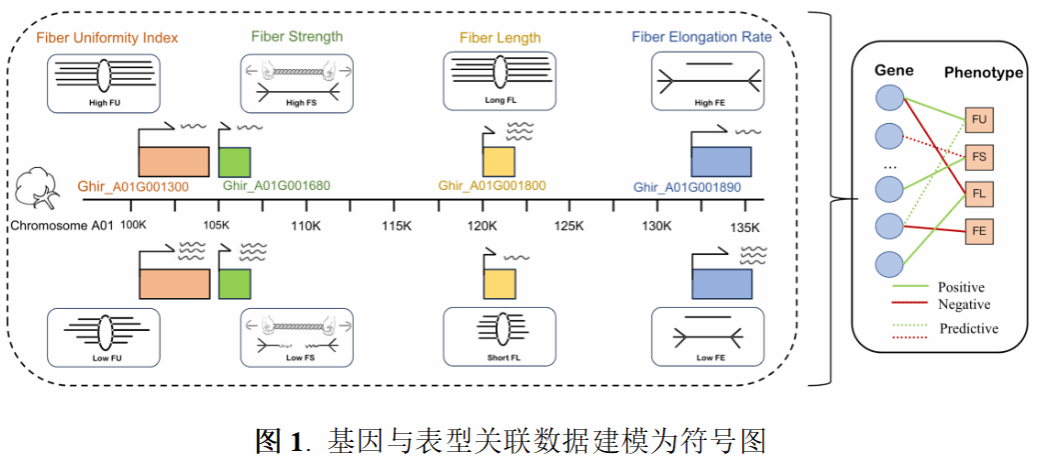
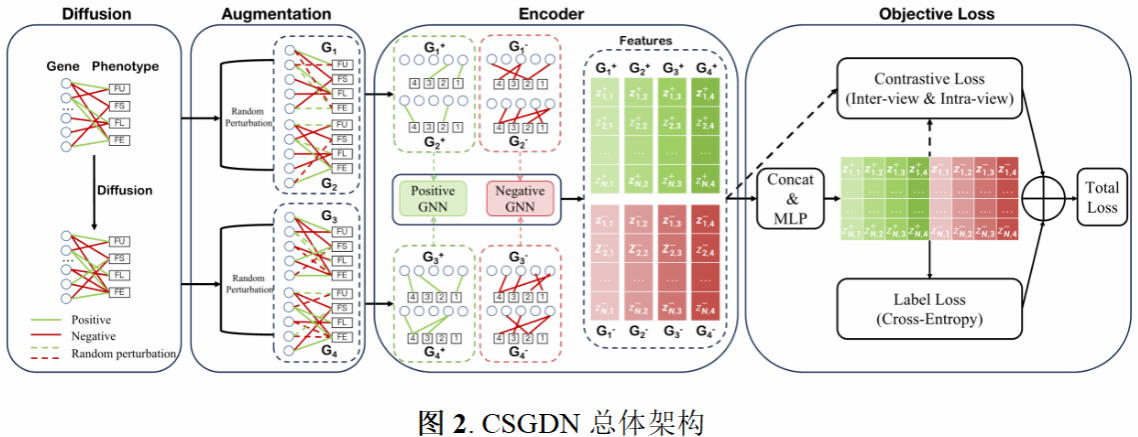
本研究主要针对传统基因-表型正负关联预测方法中的高成本和多噪音问题,首次提出一种结合图扩散的数据增强方法与对比学习方法的图神经网络框架(CSGDN)。该模型将基因-表型正负关联数据集建模为符号二部图(图1),基因与表型分别作为图的两个节点集,正/负的关联关系作为正/负边集。如CSGDN总体架构图(图2)所示,通过有符号图扩散理论,利用符号图随机游走计算结点得分并生成扩散图,从而在小数据集中挖掘隐藏关联以降低成本。同时,采用随机扰动技术对原始图和扩散图进行数据增强,生成多种增强图并分别用图神经网络(GNNs)学习正负连接表示,再通过多视图对比学习损失统一节点表示,增强模型鲁棒性以抵抗噪音干扰。
CSGDN能使用小样本数据预测物种的基因-表型关联,并在噪音环境下表现出优异抗干扰能力。棉花、油菜、小麦三个作物数据集的测试结果表明CSGDN的性能优于多个baseline模型。同时,在随机采样和随机干扰的数据集上进行测试时,CSGDN也在多个指标上优于baseline模型。最终表明CSGDN模型在基因-表型关联预测问题的性能良好,结合数据增强方法与对比学习框架有效应对了基因-表型关联预测中的高成本与噪声问题。
信息学院本科生潘伊茹、吉星宇及植科院博士生尤佳琦为论文共同第一作者,植科院王茂军教授和信息学院张泽宇副教授为共同通讯作者,张献龙院士参与该项研究。该研究得到新疆科技厅揭榜挂帅项目、国家重点研发计划项目等资助。
