8月24日,华中农业大学果蔬园艺作物种质创新与利用全国重点实验室、湖北洪山实验室、信息学院焦文标课题组在Plant Communications在线发表了题为SynDiv: An efficient tool for chromosome collinearity-based population genomics analyses 的论文。该研究开发了一个名为SynDiv的工具,可用于在大规模群体中量化评估染色体共线性多样性;并首次提出了基于基因组共线性的群体分化指数Syn-FST,有助于定量分析那些传统群体遗传学分析方法难以触及的基因组区段。SynDiv的开发将进一步促进染色体结构进化、群体遗传学和功能基因快速筛选定位等方面的研究。
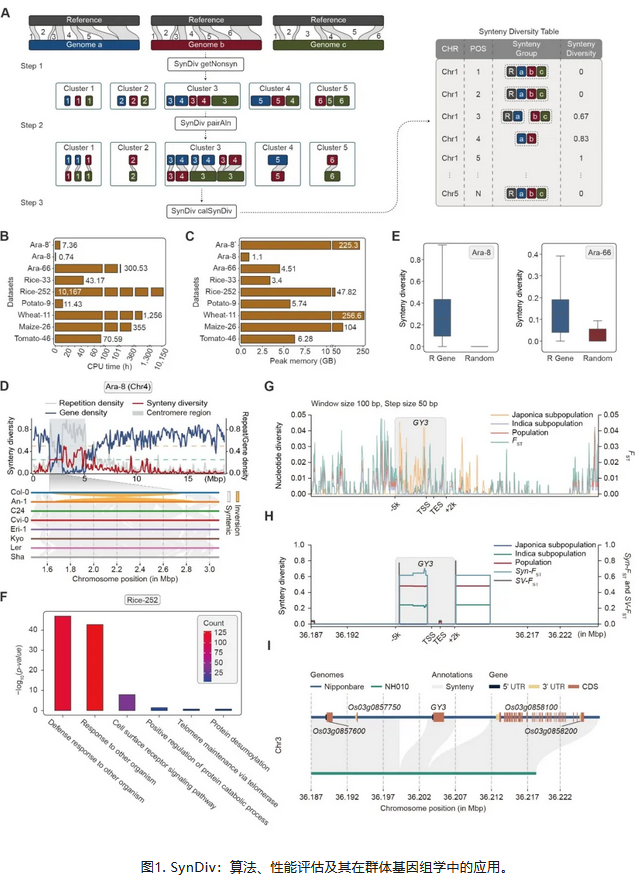
第三代长度长测序技术的迅猛发展推动了大规模染色体水平的从头组装。在此基础上,群体规模的基因组比较分析鉴定了大量结构变异(SV),其中许多SV与控制重要性状的基因位点相关。然而,基于SNP等简单标记的传统群体遗传学方法难以应对基因组中的大量结构变异区段。为了量化群体水平上基因组共线性(或非结构变异)的保守程度,焦文标课题组前期提出了一个称为共线性多样性synteny diversity(π syn)的参数 (Jiao and Schneeberger, 2020)。与核苷酸多样性这一衡量碱基差异的指标不同,π syn 量化的是染色体水平共线性的差异程度。然而,先前的计算流程较为复杂、计算资源消耗过大,难以适用于大规模群体。为了解决这些问题,作者开发了一款新的生物信息软件SynDiv,能够在计算资源友好的前提下,快速准确地衡量不同植物群体中的π syn。
为了验证SynDiv在不同植物群体中的性能,作者在拟南芥、水稻、小麦等多个物种的群体进行了测试。结果表明,相比于之前的方法,SynDiv在拟南芥群体中的内存消耗减少了200倍,运行时间缩短了90倍。此外,SynDiv能在合理计算时间和内存需要下,处理包含252个水稻基因组从头组装的大规模群体、以及包含11个小麦基因组组装序列的超大基因组群体。进一步分析共线性多样性较高的区域(结构变异相对较多),即染色体重排热点(HOT)区域,作者发现这些区域重复序列密度较高且基因密度较低,且显著富集与生物和非生物胁迫响应相关的基因,如抗病基因、一些次级代谢通路相关基因,揭示了这些高度变异区域可能参与了物种适应性的动态调控。
为深入探究群体的遗传结构和进化历史,特别针对那些没有SNP标记的区域,作者提出了一个新的群体遗传参数:基于基因组共线性多样性的群体分化指数Syn-FST。与传统群体遗传参数FST不同,Syn-FST关注的是共线性多样性而不是单个碱基的差异。在水稻群体(包括粳稻和籼稻亚群)的分析中,前1% Syn-FST较高区域的基因显著富集与非生物刺激反应相关的生物途径,这些基因可能是水稻驯化过程中的选择目标。例如,在谷物产量相关基因GY3上游(-3.9 kb ~ -0.6 kb)区域因转座子元件(TE)插入,Syn-FST值显著升高,此区域先前已通过 QTL 作图得到确定。此外,水稻产量相关的两个基因RNR10和SLB1/2也能基于Syn-FST筛选出来,且能与已有的QTL结果相吻合。
综上,SynDiv是一种快速量化基因组共线性差异及种群内外基因组结构变化的工具。结合我们提出的新参数Syn-FST,SynDiv 能够量化和表征传统群体遗传分析中常被忽视的基因组区域。更重要的是,SynDiv还可用于染色体结构进化分析、选择检测以及与性状相关的候选基因位点的精准定位。该软件及测试数据可通过Github网站免费获取 https://github.com/JiaoLab2021/SynDiv 。
华中农业大学果蔬园艺作物种质创新与利用全国重点实验室、湖北洪山实验室、信息学院博士研究生杜泽臻、贺家宝为论文共同第一作者,焦文标教授为通讯作者。该工作得到了国家自然科学基金面上项目和优秀青年科学基金项目(海外),以及重点实验室自主培育项目的资助。
参考文献:
Jiao, WB., and Schneeberger, K. (2020). Chromosome-level assemblies of multiple Arabidopsis genomes reveal hotspots of rearrangements with altered evolutionary dynamics. Nat Commun 11:989.
原文链接:
