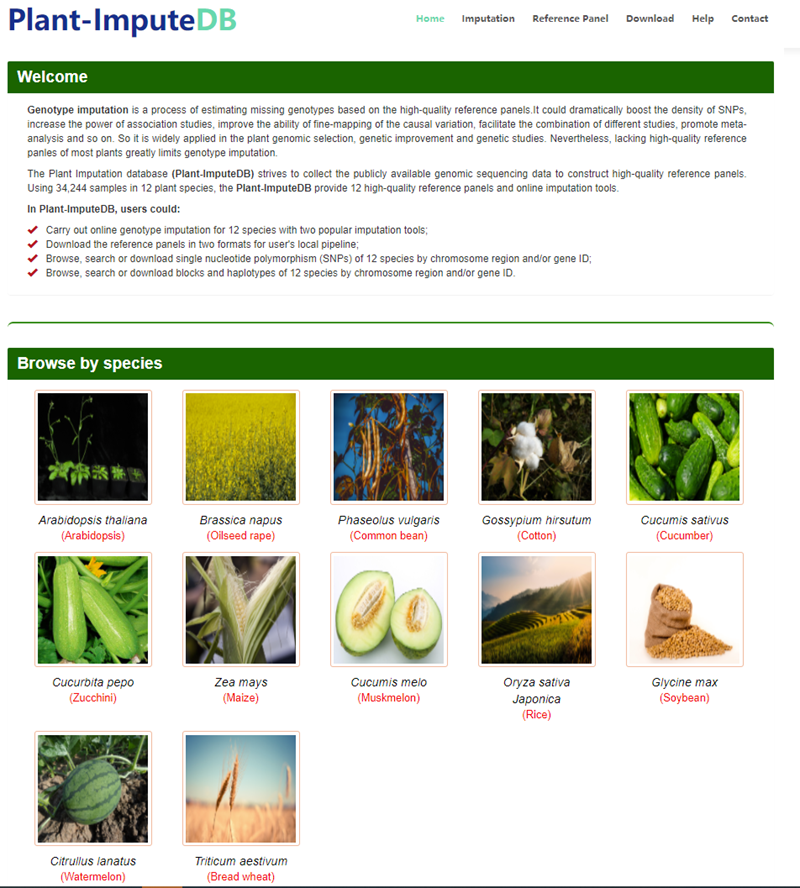
(图文|辛西)近日,华中农业大学信息学院生物信息团队在国际著名学术期刊“Nucleic Acids Research”中发表题为“Plant-ImputeDB: an integrated multiple plant reference panel database for genotype imputation”的研究成果。该研究通过收集包括水稻,玉米、小麦、油菜和棉花等12个重要农作物的遗传变异信息,构建了植物中首个多物种的高质量遗传参考变异库,为植物遗传育种研究提供了宝贵资源。
据了解,基因型填充(Genotype imputation)是根据参考面板(reference panles,又译参考集)中的单体型和基因型对目标样本中缺失基因型的估计填充过程。基因型填充可有效增加单核苷酸多态性(SNPs)密度,因此被广泛用于相对便宜和低密度的SNP芯片的填充以进行大规模全基因组关联研究(GWAS)。然而,大多数植物缺乏高质量的参考面板,这极大限制了基因型填充在植物中的应用。为克服这一限制,研究者开发了植物遗传参考变异库--Plant-ImputeDB,数据库通过整合了包括水稻,玉米、小麦、油菜和棉花等12个重要农作物的遗传变异信息和全基因组重测序数据(包括34,244个样本的约6,990万个SNP的遗传变异信息),构建了植物中首个多物种参考面板的综合数据库。该数据库提供在线的基因型填充,支持SNP和基因组block两种方式搜索、浏览并提供相应数据下载,同时接受不同类型的基因组变异数据的提交,提供对所有公开可用数据的免费和开放访问,以支持全球范围相关研究。
华中农大信息学院硕士生高英杰、博士生杨植全、杨文倩为论文并列第一作者,牛晓辉副教授、杨庆勇副教授和龚静教授为论文共同通讯作者,博士研究生杨彦波参与了研究工作,该研究成果得到中央高校基本科研业务费、国家重点研发计划、华中农大科技创新基金等项目资助。
