
MLLASSO模型的算法流程图
GPGs与GUGs的GO富集分析与TF家族富集分析
(图文|辛西 學海)近日,我校和加州大学河滨分校研究团队在基因组预测研究方面取得新进展,相关研究成果以A directed learning strategy integrating multiple omic data improves genomic prediction 为题发表在 Plant Biotechnology Journal 期刊上。
遗传学的一个主要任务是建立基因型与表型的联系。在过去的十多年中,全基因组关联分析(GWAS)成功鉴定了数百上千个与表型显著关联的遗传位点。然而这些探测到的遗传变异位点只能解释很小一部分的表型变异,所以基于它们来构建的表型预测模型具有明显的局限性。基因组预测提出使用全基因组范围的遗传变异位点构建表型预测的统计模型,在实践中展现出了明显的优势。因此,基因组预测统计模型方面的进步将会加快育种进程,该领域是当前分子育种领域研究的一个热点。
已有的基因组预测方法大多仅仅使用群体的基因型信息,通过构建GBULP、LASSO等线性模型对表型进行预测。但是,对于如产量这类多基因控制的复杂性状,目前的线性方法由于无法抓取位点之间的互作信息而导致预测能力普遍不高。而神经网络和随机森林等非线性方法的预测能力也提高不大,显示当前方法存在共性瓶颈。
近年来,各类组学技术得到了广泛应用,产生了大量的包括转录组、代谢组在内的多层次的中间组学数据。而前期的报导表明,基于中间组学数据的表型预测能提高产量性状的预测能力,说明中间组学数据在预测复杂性状方面提供了额外的信息。但是,一个明显的限制是获取一个群体的转录组、代谢组数据既耗时也耗力。
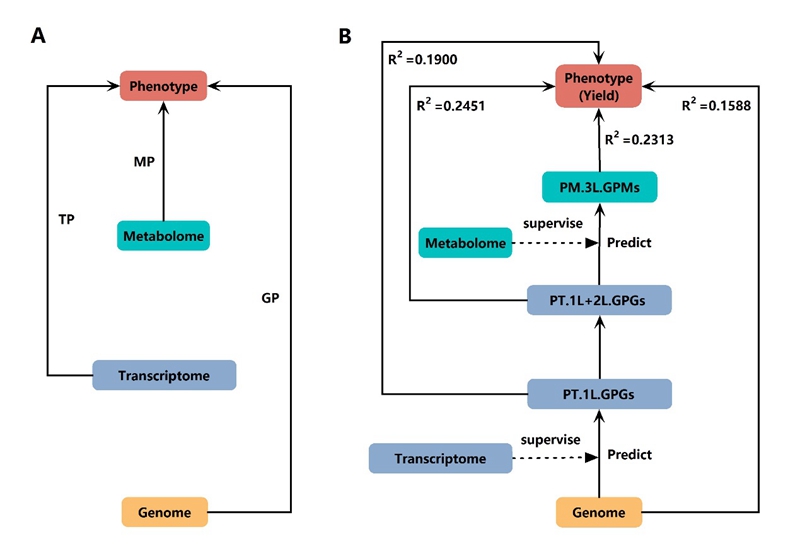
该研究提出一个新颖的整合多组学数据的基因组预测模型MLLASSO。MLLASSO通过构建多层LASSO模型,一步步地依次将基因组、转录组、代谢组信息有机整合在一个模型中。在一个水稻重组自交系群体的测试中,该模型成功地将产量的预测能力从单层仅基于基因型信息的LASSO模型的0.1588提高到0.2451,预测能力提高了超过50%。该模型的最大优势是,基于中间组学数据监督下的有向学习建立模型,而模型建立后,仅仅需要个体的基因型作为输入,这一点大大提高了其在植物育种方面的实用性。
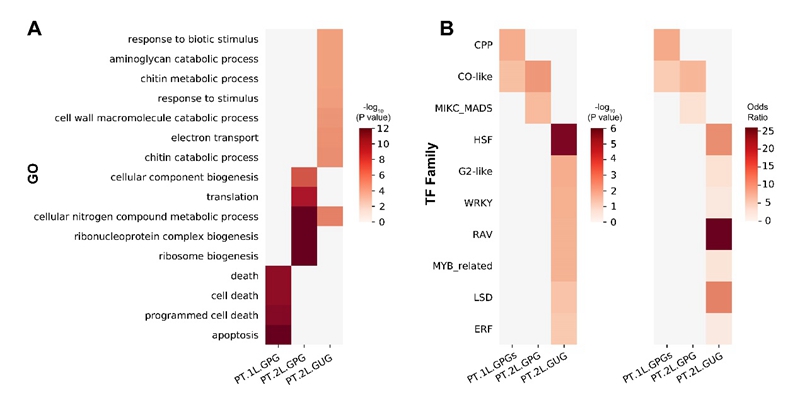
在预测过程中,一个发挥重要作用的关键点是遗传可预测基因(genetically predictable genes, GPGs)。GPGs的表达量可以被准确预测,而更多的GPGs会提高产量的预测能力。有意思的是,结合前期报导的eQTL信息、GO富集分析和转录因子统计,该研究发现,最先可由基因型准确预测的基因(PT.1LGPGs)大部分具有cis-eQTL,而基于基因型和PT.1LGPGs可准确预测另一些基因的表达量,这些基因(PT.2LGPGs)大多具有trans-eQTL。而遗传不可预测基因(genetically unpredictable genes, GUGs)富含转录因子和响应环境刺激的基因。通过对水稻56个TF家族的富集分析,该研究发现GPGs中显著富集了两个产量相关的CO-like家族和MIKC_MADS家族。其中,CO-like家族中包含了一个控制每穗粒数、株高和抽穗期等多个产量相关性状的主效基因---Ghd7[5]。这些发现都为模型的可解释性提供了有力支撑。
最后,该研究指出,MLLASSO模型的成功之处在于他们开发了基于中间组学数据监督下的有向学习策略。在中间组学的监督下,MLLASSO模型更加智能地组合相关遗传位点成为模块,更能抓取基因间的互作信息。
该研究由信息学院胡学海副教授、谢为博教授与加州大学河滨分校徐士忠教授合作完成,胡学海副教授为该文第一作者,徐士忠教授为该文通讯作者。该项目得到华中农大自主创新基金、国家重点研发计划、国家自然科学基金等项目资助。
[1]. Hu, X., Xie, W., Wu, C., and Xu, S. (2019) A directed learning strategy integrating multiple omic data improves genomic prediction. Plant Biotechnol. J., https://doi.org/10.1111/pbi.13117.
[2]. Xu, S., Xu, Y., Gong, L. and Zhang, Q. (2016) Metabolomic prediction of yield in hybrid rice. Plant J. 88, 219–227.
[3]. Hua, J., Xing, Y., Wu, W., Xu, C., Sun, X., Yu, S. and Zhang, Q. (2003) Single locus heterotic effects and dominance by dominance interactions can adequately explain the genetic basis of heterosis in an elite rice hybrid. Proc. Natl Acad. Sci. 100, 2574–2579.
[4]. Wang, J., Yu, H., Weng, X., Xie, W., Xu, C., Li, X., Xiao, J. et al. (2014) An expression quantitative trait loci-guided co-expression analysis for constructing regulatory network using a rice recombinant inbred line population. J. Exp. Bot. 65, 1069–1079.
[5]. Xue, W., Xing, Y., Weng, X., Zhao, Y., Tang, W., Wang, L., Zhou, H. et al. (2008) Natural variation in Ghd7 is an important regulator of heading date and yield potential in rice. Nat. Genet. 40, 761.
