南湖新闻网讯(通讯员 曹志林)近日,华中农业大学作物遗传改良国家重点实验室李兴旺教授和李国亮教授团队合作绘制了水稻活跃基因以及异染色质参与的高分辨率三维基因组图谱,揭示了水稻三维基因组结构对基因的转录调控,以及遗传变异对三维基因组结构及基因表达的影响。2019年8月13日,该研究成果以“Chromatin loops associated with active genes and heterochromatin shape rice genome architecture for transcriptional regulation”为题在线发表在国际学术期刊《Nature Communications》。
近年来,利用传统的Hi-C技术,水稻的三维基因组研究取得了多项重要发现,鉴定了一些较大的结构单元,如区室结构(compartment),拓扑结构域(TAD)等。然而,受限于分辨率不高等因素,Hi-C方法很难精确地检测到基因启动子-启动子交互(Promoter-promoter Interaction, PPI)、染色质环(chromatin loop)等精细的三维基因组结构。
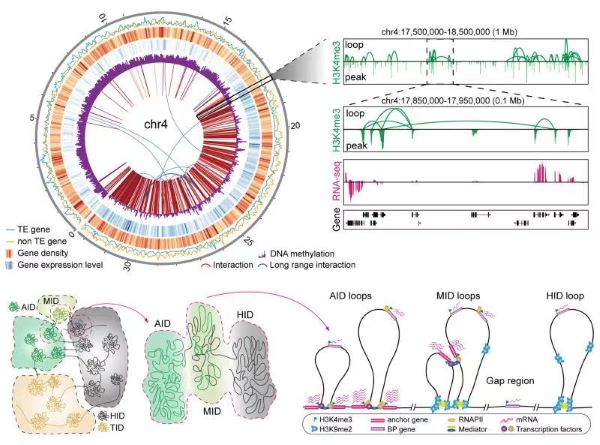
本研究利用改进的Long-read ChIA-PET技术,构建了RNA聚合酶II (RNAPII)、组蛋白修饰H3K4me3介导的水稻活跃基因的染色质交互图谱以及组蛋白修饰H3K9me2介导的异染色质交互图谱,并进一步系统地分析了三种交互的基本特征。结果显示,RNAPII介导了28,213个染色质远程互作,H3K4me3修饰区域(主要是启动子区)参与了11,230个远程互作,H3K9me2修饰区域(主要是异染色质区)参与了11,590个远程互作。分析表明,与未参与染色质交互的结合位点比较,参与PPI交互的结合位点覆盖了更高活性和广泛表达的基因,且PPI锚定基因倾向于协同表达,而异染色质交互的染色质环形成了水稻染色体构象中相对稳定的结构模块。基于此,研究人员提出水稻基因组在空间上可划分为几类具有不同转录活性的染色质交互模块(CID),共覆盖了大约82%的水稻线性基因组区域。
此外,通过水稻不同品系间基因组变异数据的分析,研究人员揭示了功能性遗传变异对水稻染色质拓扑结构及基因转录调控的影响。同时通过整合已发表的eQTL数据,提出了染色质空间邻近为eQTLs-etraits遗传关系提供了三维基础。这个成果的水稻高分辨率三维基因组学研究和基因间的互作调控信息,有助于我们深入理解水稻空间结构与各种表观修饰及基因表达的关系,为水稻遗传改良和其他经济作物的研究提供重要的指导意义和科学价值。
值得注意的是,本研究是继2019年6月14日在《Nature Communications》发表玉米高分辨率三维基因组图谱后,该团队在植物三维基因组研究领域的又一重要进展。
华中农业大学博士后赵伦、硕士研究生王双琦和博士研究生曹志林为论文共同第一作者。生命科学技术学院李兴旺教授和信息学院李国亮教授为共同通讯作者。研究得到了国家重点研发计划、国家自然科学基金,中国博士后科学基金、中央高校基本科研专项资金以及作物遗传改良国家重点实验室自主课题等项目和基因组研究与水稻遗传改良创新团队的支持。