
南湖新闻网讯(通讯员 肖琴)近日,我校作物遗传改良国家重点实验室李兴旺和李国亮教授研究团队研究成果以“The landscape of promoter-centered RNA-DNA interactions in rice”为题在Nature Plants发表。研究开发了RNA-DNA交互技术(ChRD-PET),绘制了水稻染色质结合RNA与启动子的全基因组互作图谱, 揭示了R-loop中RNA种类多元性与来源多源性,并报道了RNA在染色质环和染色质拓扑结构域等不同层级结构中的交互特征。
真核生物的染色质在细胞核内折叠包装形成了复杂而有序的三维空间结构,参与基因转录等多种生命活动。其中,DNA、RNA和蛋白质彼此间会形成复杂的互作网络,但染色质结合RNA在水稻三维基因组结构中的功能尚不清楚。
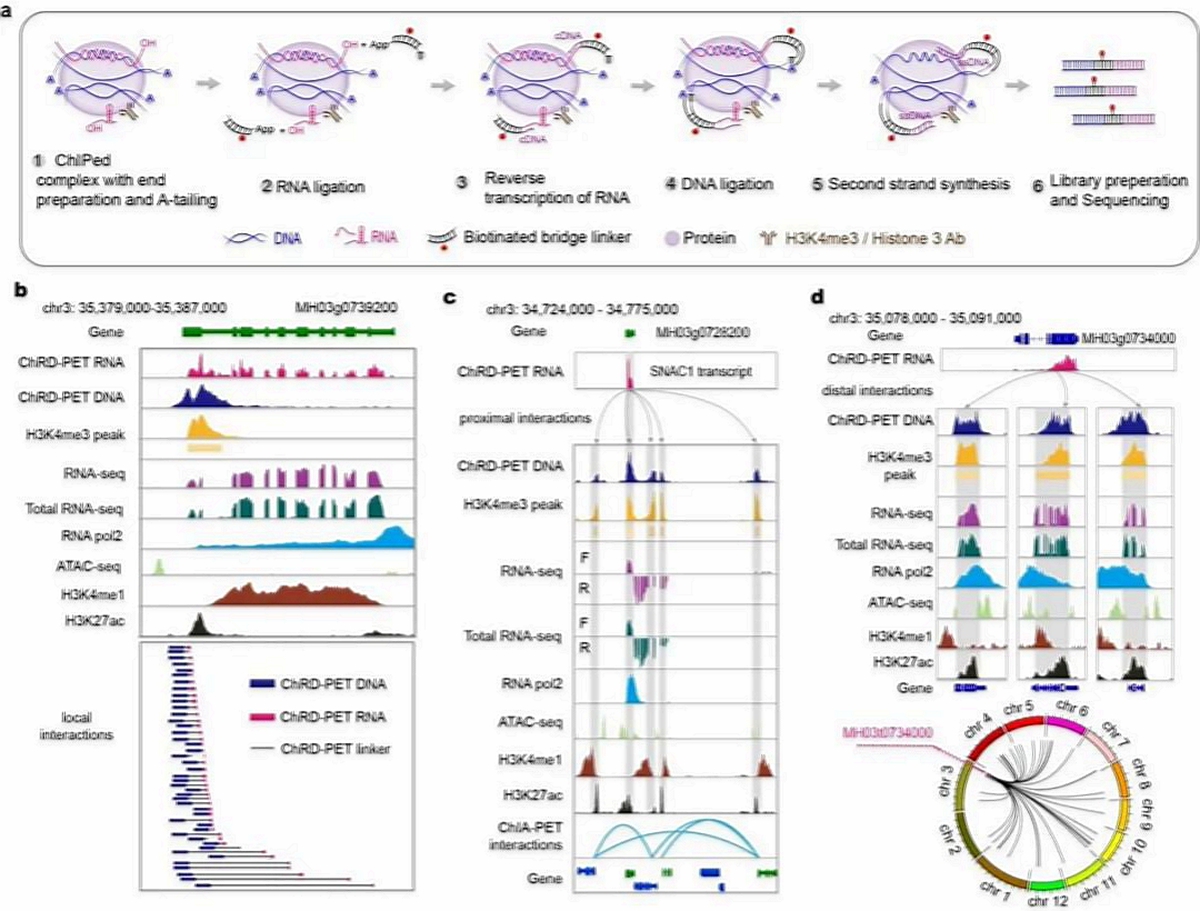
研究首先报道了一种植物RNA-DNA互作研究技术——ChRD-PET(Chromatin-associated RNA-DNA interactions followed by paired-end-tag sequencing), 其主要原理是利用特异组蛋白修饰抗体,通过染色质免疫共沉淀富集DNA-RNA-蛋白质复合物,用一种生物素标记的DNA bridge linker,将复合物上空间接近的RNA和DNA进行连接,通过高通量测序获得DNA和RNA交互的数据。利用这一技术,研究人员构建了水稻H3 ChRD-PET和H3K4me3 ChRD-PET数据,分别表征染色质结合RNA在全基因组上的结合位点,以及染色质结合RNA与H3K4me3修饰区域(标记水稻活跃启动子)的互作图谱。进一步系统地分析了RNA与染色质交互的基本特征。
结果显示,有的RNA在转录原位参与交互(原位交互,35.9%),有的与附近DNA形成交互(邻近交互,8.8%),有的则跨染色体去参与交互(远程交互,55.3%)。在启动子区域,编码RNA和非编码RNA,参与了广泛的RNA-DNA交互,暗示了非编码RNA在基因启动子区对基因转录调控的潜在功能。
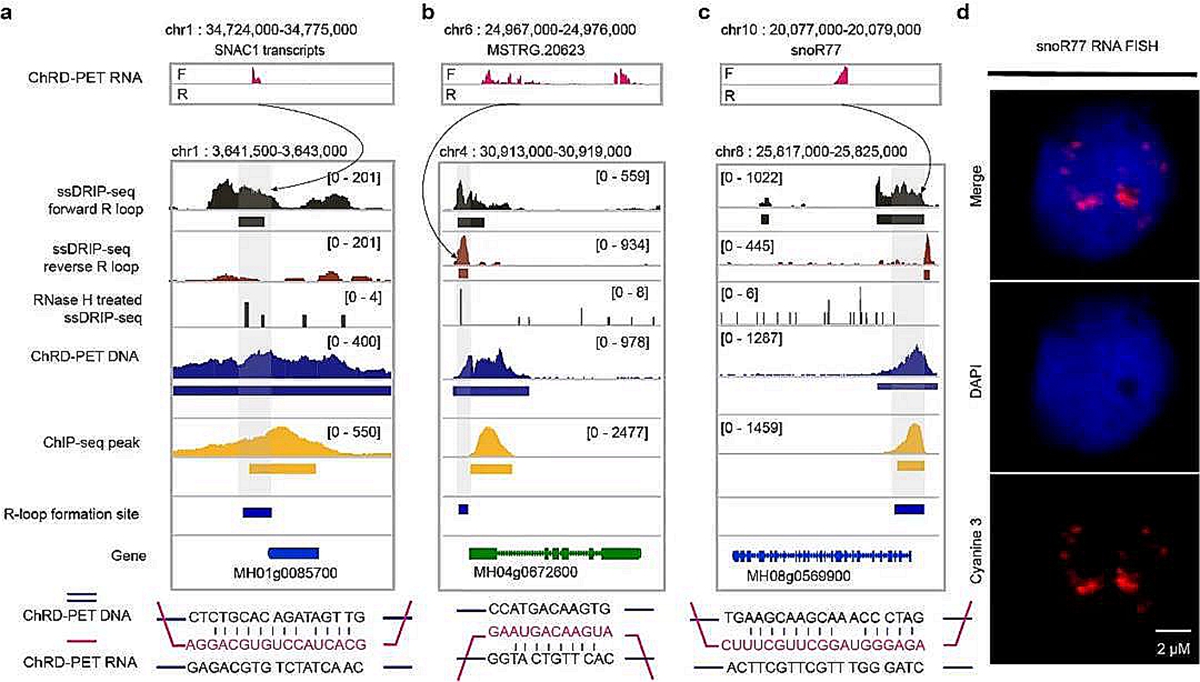
一部分染色质结合RNA可以与DNA单链形成RNA-DNA hybrid(R-loop),通过检测RNA-DNA hybrid中DNA链可以反映R-loop在基因组中的形成位置,而R-loop中RNA链的来源和组成尚不清楚。为了探究这一问题,研究人员将ssDRIP-seq数据和H3K4me3 ChRD-PET数据联合分析,鉴定了8562个R-loop衍生的RNA-DNA交互,发现除编码RNA外,非编码RNA可以通过远程交互的方式参与R-loop的形成,说明了R-loop中RNA的多样性和多源性,这一发现使人们对R-loop的组成和形成有了更深的认识。
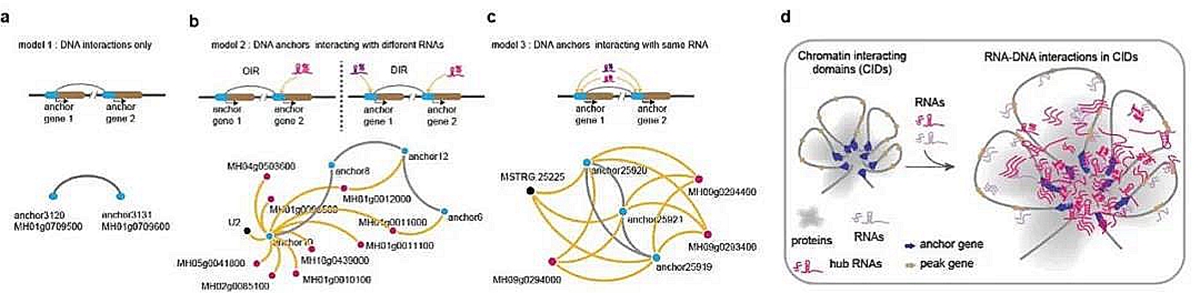
此外,通过与水稻表观基因组数据联合分析,研究人员揭示了预测增强子可以招募更多的RNA来交互,同时本身也转录更多的RNA去参与交互。进一步与水稻的三维基因组H3K4me3 ChIA-PET数据整合分析,发现RNA与染色质环存在三种交互模式:染色质环的gene上没有RNA交互、染色质环的gene上有不同的RNA交互、染色质环的gene和同一RNA交互。此外,在水稻染色质交互的拓扑结构中,一个基因转录的RNA可以和这个结构中一半以上的DNA有交互,共鉴定了614个这样的拓扑结构域 。
综上所述,该研究开发了RNA-DNA交互技术,绘制了启动子附近的RNA-DNA交互图谱,并分别分析了DNA序列层面、一维线性表观修饰和三维染色质结构层面的RNA-DNA交互特征,推测RNA在染色质不同层级的三维结构中可能发挥着重要作用,有助于深入理解RNA与水稻染色质空间结构、各种表观修饰及基因转录调控的关系,为水稻RNA功能研究、遗传改良和其他经济作物的研究提供重要的研究资源和科学价值。
值得一提的是,该研究是继一系列水稻、玉米高分辨率三维基因组图谱发表后,该团队在植物三维基因组研究领域的又一重要进展。
华中农业大学博士研究生肖琴、博士研究生黄星宇为论文共同第一作者,李兴旺教授、李国亮教授为论文共同通讯作者。清华大学孙前文教授、中国农业科学院(深圳)农业基因组所徐炜研究员参与了相关研究。该研究得到了国家自然科学基金,中国博士后科学基金、中央高校基本科研专项资金以及作物遗传改良国家重点实验室自主课题等项目基金的支持。
审核人:李兴旺
【英文摘要】
Chromatin-associated RNAs play key roles in various biological processes. However, both their repository and conjugation genomic loci and potential functions remain largely unclear. Here, we develop an effective method for mapping of chromatin-associated RNA–DNA interactions, followed by paired-end-tag sequencing (ChRD–PET) in rice. We present a comprehensive interaction map between RNAs and H3K4me3-marked regions based on H3K4me3 ChRD–PET data, showing three types of RNA–DNA interactions—local, proximal and distal. We further characterize the origin and composition of the RNA strand in R-loop RNA–DNA hybrids and identify that extensive cis and trans RNAs, including trans-non-coding RNAs, are prevalently involved in the R-loop. Integrative analysis of rice epigenome and three-dimensional genome data suggests that both coding and non-coding RNAs engage extensively in the formation of chromatin loops and chromatin-interacting domains. In summary, ChRD–PET is an efficient method for studying the features of RNA–chromatin interactions, and the resulting datasets constitute a valuable resource for the study of RNAs and their biological functions.
论文链接:https://www.nature.com/articles/s41477-021-01089-4
