(图文|王子林 审核人|李立)11月26日上午,学院“Happy hour”2021年第22期交流会在逸夫楼C603会议室举行,腾讯会议同步直播。中科院基因所张治华研究员进行了单细胞三维基因组学相关的学术汇报,学院多位师生参与了线上和线下的交流活动,副院长李国亮教授主持交流会。
张治华研究员首先介绍了三维基因组学的背景以及发展现状,并指出拓扑相关结构域(TADs)在细胞三维结构中的作用,以及在单细胞尺度上也存在类似的TAD结构(TLD)。
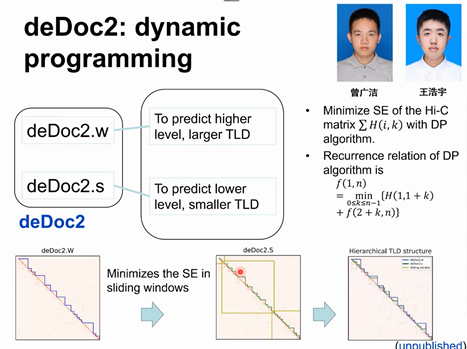
张治华就之前和现有工作进行了详细介绍:首先是DeDoc,一种基于信息熵理论和贪婪算法来寻找局部最优的TAD结构,但这个算法在单细胞数据中效果并不理想;其次是deTOKI,一种基于滑动窗口和非负矩阵分解的优化算法,从Hi-C矩阵中预测TAD边界,并在抽样、仿真和单细胞Hi-C数据中都达到较好效果,使用deTOKI能鉴定到TAD边界在单细胞中的变异,并且揭示了TAD在不同细胞之间存在一定异质特征;最后是deDoc2,在deDoc基础上进行了优化,使用动态规划算法寻找全局最优解,并且在大窗口和小窗口等多层次上进行TAD边界分析,结果显示deDoc2在各种不同数据中均表现良好,且对数据填充不敏感,因该算法利用了数据的全局特征,且有较强鲁棒性。
随后张治华介绍了猪的胚胎三维基因组的研究结果,通过人工授精、孤雌和孤雄的胚胎干细胞实验,他们发现猪胚胎发育早期中存在染色质凝聚,在人工授精胚胎细胞的四细胞时期的样本中开始出现一个早期superdomain直到发育末期,且在孤雌和孤雄样本中存在decompartmentlalization现象并在桑葚胚时期重建。
张治华最后展示了他们的数据可视化软件Delta和Delta.AR,能对三维基因组数据进行基因组结构、拓扑结构和物理三维结构可视化,还向我们展示了物理结构AR可视化,通过AR设备可近距离观察基因组的三维结构和结合信息。
报告结束,参会师生进行了积极交流和深入研讨。李立教授对这些TAD研究十分感兴趣,探讨了如何研究单细胞中TAD的特征,李国亮教授就测序深度和单细胞TAD的变化情况进行了积极交流,还有学生深入探讨了单细胞数据分型的验证标准及效果评估,大家充分交流想法,思维碰撞,收获颇丰,本次交流会在愉快氛围中结束。
