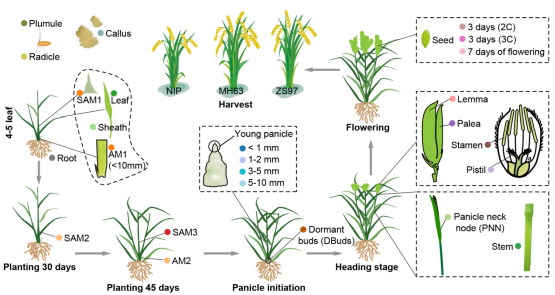
图1水稻全生育期调控组图谱的取样组织和时间点
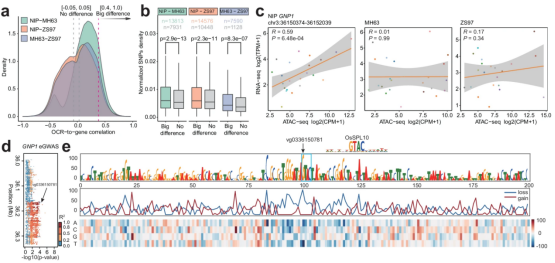
图2 利用调控组深度学习模型鉴定关键变异位点
(图文|辛西 编辑|信息 审核|谢为博)近日,我校信息学院谢为博课题组与南京大学陈迪俊课题组在Nature Communications杂志上联合发表了题为“Comprehensive mapping and modelling of the rice regulome landscape unveils the regulatory architecture underlying complex traits”的研究论文。
该研究利用谢为博教授课题组之前开发的UMI-ATAC-seq技术,绘制了迄今最全面的水稻调控组图谱,涵盖了3个代表性水稻品种全生育期的23个组织。研究发现,水稻基因组中约15.6%的区域可能具有调控功能,这一比例远高于以往植物研究中的报道。研究进一步鉴定了一系列时空特异的增强子,并通过联合转录组数据,建立了5.9万个调控区和靶基因的关联。利用这一资源,研究团队重新评估了已发表的水稻全基因组关联分析研究的结果,鉴定并验证了影响种子萌发率的新基因OsbZIP06。此外,研究还优化了深度学习模型来解码水稻调控组的调控语法,实现了对组织特异性染色质可及性的有效建模。研究人员利用所构建的模型可从基因组序列预测不同品种间的调控动态,揭示了品种间顺式调控分化和性状差异的遗传基础。总之,该研究不仅增强了我们对水稻调控元件的理解,也为针对调控区的基因编辑和育种策略提供了重要资源。
信息学院已毕业硕士(现为南京大学博士生)祝涛和我校夏春皎博士为该论文的共同第一作者,我院校友南京大学生命科学学院陈迪俊副教授和我院谢为博教授为该论文的共同通讯作者,该研究得到国家科技创新2030重大项目、国家自然科学基金、南京大学登峰人才支持计划、湖北省自然科学基金、中央高校基本科研业务费专项资金和洪山实验室的资助。
论文链接:https://www.nature.com/articles/s41467-024-50787-y
