(图文|九九|兽医研圈)抗生素耐药性已成为全球公共卫生领域一大挑战。特别是肠外致病性大肠埃希菌(Extraintestinal Pathogenic Escherichia coli, ExPEC),它们不仅在人类中引起严重泌尿道感染、血流感染、新生儿脑膜炎等,且在动物中也造成显著健康和经济损失。尽管ExPEC对人类和动物健康构成的威胁日益增加,但人们对动物源ExPEC多样性、抗生素耐药性及其与人类感染的联系了解甚少。特别是在中国,作为世界上最大猪肉生产和消费国,猪源ExPEC的种群结构和耐药性特征对于理解其在公共卫生中的作用至关重要。然而目前对猪源ExPEC的系统研究相对缺乏,对其耐药基因(antibiotic resistance genes, ARGs)的传播机制和潜在公共卫生影响的认识也不够全面。因此,本研究通过对来自中国499株猪源ExPEC进行大规模基因组测序和抗生素耐药性测试,旨在揭示其种群结构、耐药性特征以及与人类病原体的关联性,为制定有效的监控和控制策略提供科学依据。
7月10日,华中农大郑金水&谭臣团队在nature communications 上发表题为“Population structure and antibioticresistance of swine extraintestinal pathogenic Escherichia coli from China”的研究论文,该研究探索了 ExPEC 的群体结构、耐药性表型与基因型之间的关系及这些 ExPEC 对人类感染的假定贡献。
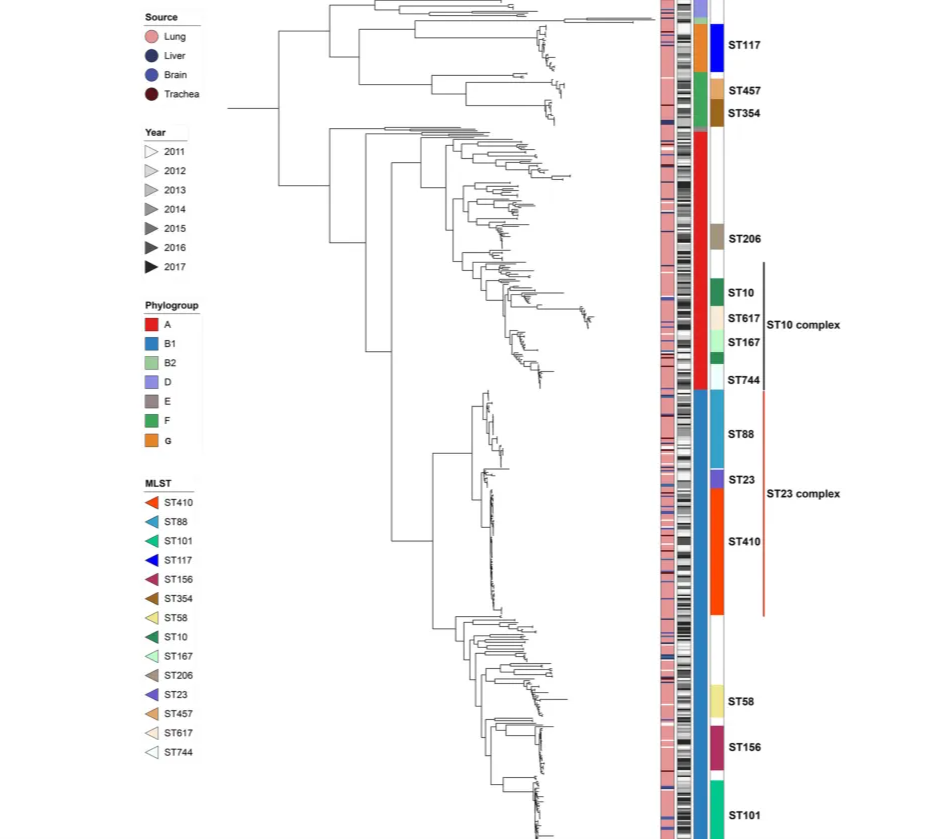
来自中国的猪源肠外致病性大肠埃希菌(ExPEC)表现出高度多样化的种群结构。通过大规模基因组测序分析,研究揭示了这些细菌在系统发育上具有显著的多样性,其中超过80%属于B1和A进化群。此外,还发现与人类来源大肠杆菌菌株具有遗传相关性的15个猪源ExPEC菌株,表明它们可能在人畜共患病中的潜在角色。这些发现强调了监测和控制动物源ExPEC感染的重要性,因为它们可能成为人类健康影响的耐药基因库。
在中国猪源肠外致病性大肠埃希菌(ExPEC)中,与肠外感染相关的毒力因子编码基因非常普遍。研究发现,这些毒力因子在ExPEC菌株中广泛分布,包括粘附素、铁摄取系统、血清抵抗基因和侵袭素等。特别是,49株菌株携带了典型肠致病性大肠杆菌(EPEC)毒素基因,如志贺毒素(STX)、热敏性(LT)和热稳定性(ST)肠毒素、粘附素(如F4、F5、F18和F41)。这些发现表明,一些猪源ExPEC菌株可能代表了混合型病原类型,增加了它们在宿主体内的致病潜力。
在中国,抗生素在禽类和牲畜养殖业中被广泛使用,导致食品动物中的大肠杆菌发展出极端抗生素抗性。研究显示,485株大肠杆菌中有97%表现出多重耐药性,其中49.3%对所有七类抗生素均表现出耐药性。特别是,95.5%的菌株对四环素和氨苄西林耐药,而仅0.8%对美罗培南、亚胺培南和他唑巴坦哌拉西林耐药。此外,70%的菌株对庆大霉素耐药,15.5%对阿米卡星耐药。研究还发现,不同谱系菌株在某些抗生素的耐药性上存在显著差异。
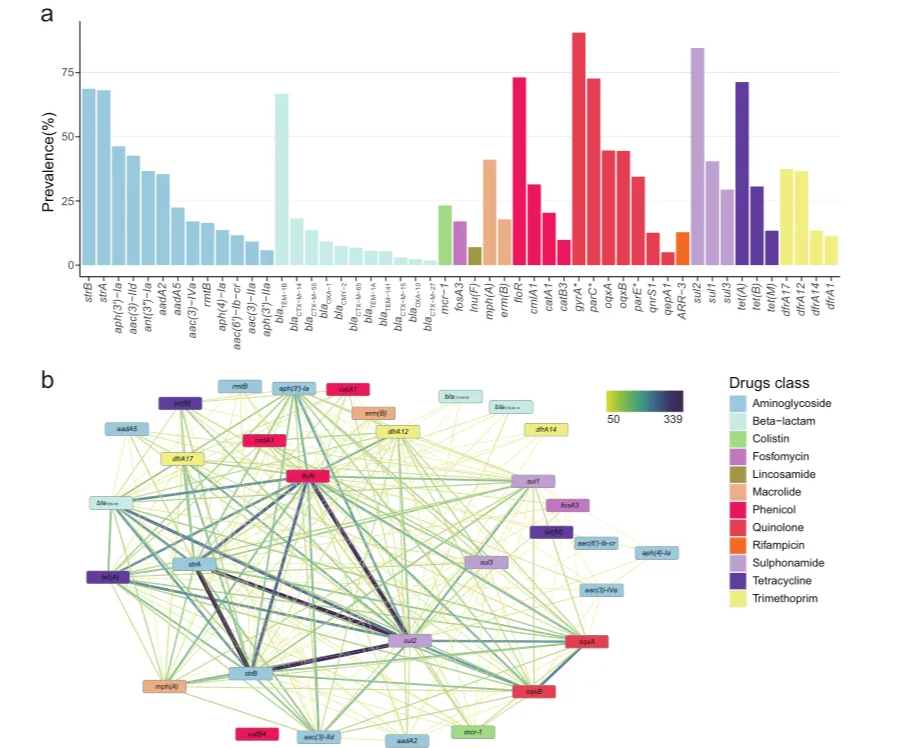
研究揭示了猪源性大肠杆菌(ExPEC)基因组中存在96种抗生素抗性基因(ARGs)和三个染色体基因(gyrA、parC和parE)的点突变,这些基因和突变与12类抗生素的抗性有关。所有基因组至少含有一种ARGs。氨基糖苷类抗性基因中,strA和strB在68%的菌株中存在,磺胺类抗性基因sul2在85%的菌株中发现。四环素抗性基因tet(A)在超过70%的菌株中存在。氟喹诺酮类抗性基因oqxA和oqxB在约45%的菌株中发现。β-内酰胺酶基因中,blaTEM-1B在67%以上的菌株中存在。此外,还发现了对粘菌素(mcr-1)和磷酸霉素(fosA3)的抗性基因。研究还比较了不同序列类型(STs)的ARGs流行率,发现ST410中某些ARGs的流行率较高,这可能是ST410流行的重要遗传因素。
研究揭示了多重耐药(MDR)和广泛耐药(XDR)大肠杆菌的遗传决定因素。通过对492个基因组的分析,发现了406种不同的抗生素抗性基因(ARGs)组合,其中96%以上仅存在于一个或两个基因组中。这些组合平均包含13个ARGs,最多可达28个。超过三分之二的组合在超过70%的基因组中发现,表明这些基因组合与MDR表型高度相关。共现网络分析显示,sul2、floR、strA和strB构成了网络的核心,与其他ARGs有联系。这些基因的存在可能导致对氨基糖苷类、氯霉素和磺胺类药物的耐药性。同时,研究还发现β-内酰胺类、四环素类和氟喹诺酮类药物的抗性基因与针对人类药物的抗性基因(如blaCTX-M-55、blaCTX-M-14、fosA3和mcr-1)共存,这可能会加剧人类药物抗性基因的传播。此外,尽管mcr-3在少数基因组中检测到,但其与mcr-1的共现也值得关注。
研究显示,抗生素抗性基因(ARGs)不仅存在于细菌的染色体上,还广泛存在于质粒中。这些基因通过染色体和质粒在细菌种群中传播,导致抗生素抗性在细菌中的快速扩散。染色体上的抗性基因通常是通过突变或重组获得的,而质粒则可以通过水平基因转移在不同细菌之间传递抗性基因。这种双重机制的存在使得抗生素抗性在细菌中的传播更加复杂和难以控制。研究还发现,某些质粒携带的抗性基因与特定抗生素的耐药性密切相关,这进一步强调了在抗生素使用和细菌管理中需要考虑这些遗传元件的作用。
研究揭示了猪源性大肠杆菌(ExPEC)携带的质粒在抗生素抗性基因(ARGs)的水平基因转移中扮演关键角色。通过比较ExPEC中的质粒与来自其他宿主(特别是人类)细菌中的质粒,发现它们之间存在显著的相似性。例如,某些质粒携带的抗性基因如blaNDM-1和blaNDM-5与人类病原体中的质粒高度相似。这些质粒不仅在ExPEC中传播,还可能在不同细菌种之间转移,包括粘菌素抗性基因mcr-1。这些发现表明,猪源性ExPEC的质粒可能通过水平基因转移将抗性基因传递给人类病原体,增加了抗生素耐药性的复杂性和挑战性。研究还发现,这些质粒携带的抗性基因不仅针对兽医药物,也针对人类治疗中重要的抗生素,进一步强调了在抗生素管理中需要考虑质粒的作用。
综上,研究总结如下3点。第一,针对抗生素抗性基因的传播机制。研究揭示了猪源性大肠杆菌(ExPEC)中抗生素抗性基因(ARGs)的传播机制,特别是通过质粒介导的水平基因转移。这些质粒不仅在ExPEC内部传播,还可能将抗性基因传递给人类病原体,增加了抗生素耐药性的复杂性和挑战性。第二,针对质粒的多样性和共存。研究发现,ExPEC携带的质粒具有高度的多样性和复杂性,它们携带多种ARGs,包括对人类治疗至关重要的抗生素的抗性基因。这些质粒的共存和传播不仅加剧了抗生素抗性的发展,也对公共卫生构成了严重威胁。第三,针对抗生素管理的重要性。鉴于质粒在抗生素抗性基因传播中的关键作用,研究结果强调了在动物饲养中合理使用抗生素的重要性。同时,加强抗生素抗性监测和管理,减少抗生素滥用,是控制抗生素抗性扩散、保护人类健康的必要措施。此外,需要进一步研究质粒的传播机制和抗性基因的共存模式,以制定更有效的抗生素管理策略。
