人类的疾病和动植物的表型性状,都与该物种的基因正确表达密切相关。而基因的表达,不完全由基因的DNA序列决定,而是同时与DNA调控元件的调控息息相关。2003年提出并实施的人类ENCODE计划(DNA调控元件百科全书计划)通过整合DNA、RNA和表观修饰等多个层面的数据建立了多组学的人类基因组DNA调控元件数据库,注释了人类基因组中数以百万计的DNA调控元件,增强了我们对人类功能基因组的理解。同时,ENCODE计划的一系列技术和成果,为后续人类或模式生物基因调控的功能挖掘提供了极大的帮助和支持。
水稻(Oryza sativa L.)是我国乃至世界重要的粮食作物,同时也是基础研究的重要模式植物。水稻基因组DNA顺式调控元件的注释和鉴定,对理解水稻基因表达调控的机理有重要意义。因此,一个整合了水稻多元表观基因组数据的数据库,将极大地方便研究人员查询和分析水稻的表观遗传信息,促进水稻表观和三维基因组研究。

https://doi.org/10.1016/j.molp.2021.08.018
8月27日,Molecular Plant 在线发表华中农业大学李国亮教授和李兴旺教授团队题为 RiceENCODE: a comprehensive epigenomic database as rice Encyclopedia of DNA Elements 的研究论文,报道了一个整合水稻多元表观基因组数据的数据库。该数据库基于团队此前发表的水稻20个品系参考表观基因组图谱(https://www.nature.com/articles/s41467-020-16457-5)和水稻高分辨率三维基因组结构数据 (https://www.nature.com/articles/s41467-019-11535-9),收集了公开发表的水稻多品种多组织多维度的表观基因组信息,展示了水稻不同类型的染色质调控元件,立体地呈现了水稻品种和组织间复杂的基因表达调控关系。这是参照人类的ENCODE计划,推进水稻功能基因组研究的重要一步。
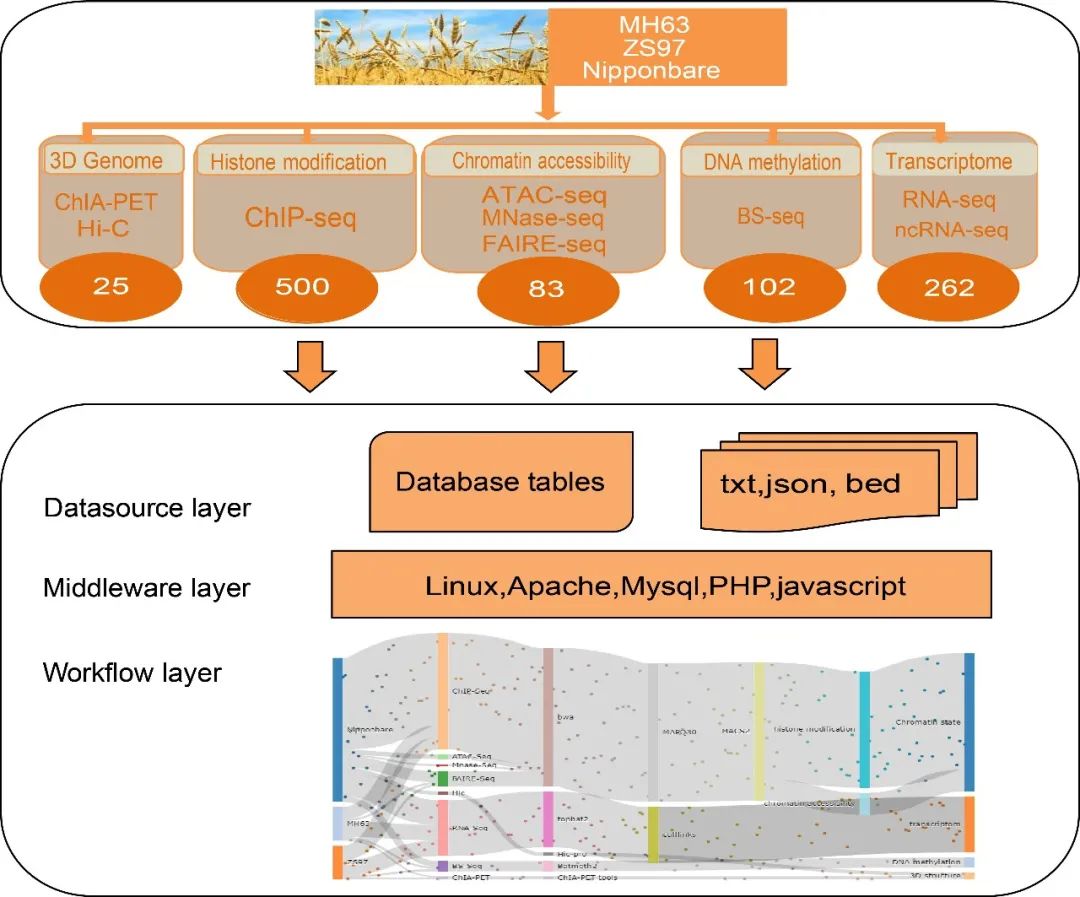
图1. 水稻DNA调控元件数据库RiceENCODE的框架结构
该数据库收集了包括ChIP-seq, ATAC-seq, MNase-seq, FAIRE-seq, BS-seq, RNA-seq, ncRNA-seq, Hi-C和ChIA-PET 等共计972套水稻高通量组学数据,通过标准化的数据处理流程,得到了多维度的高质量表观和三维基因组数据(参见图1)。
研究者构建了综合的数据库搜索页面,用户可以在数据库的基因组浏览器中查看不同品种、多种组织的表观基因组数据。用户可以根据自己的需求,选择不同类型的表观基因组数据,查询目标区域或者目标基因的表观修饰信息。该数据库还提供了大量的结果数据信息,这些数据文件都可以在下载页面下载。用户可以根据自己的需要进行下游分析。
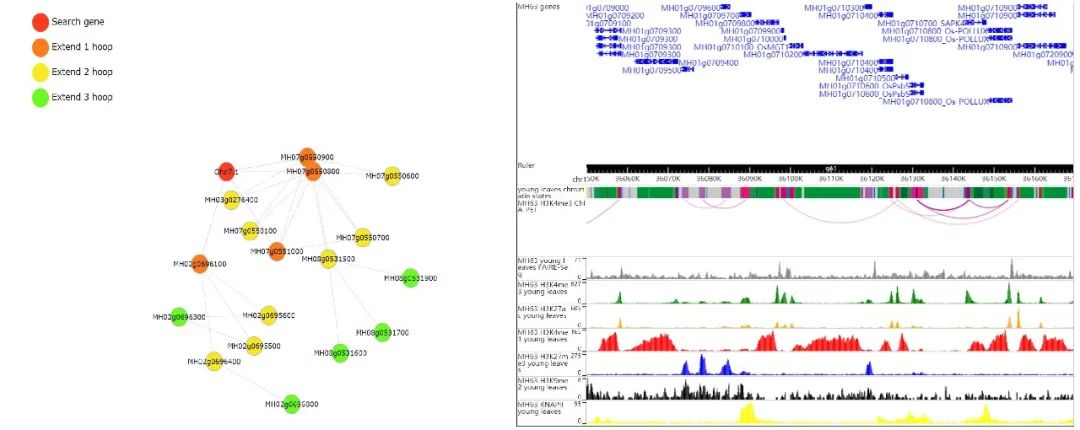
图2. 水稻的染色质交互远程网络和浏览器截图
另外,该数据库引入了水稻的三维基因组数据。用户不仅可以查询目标区间参与的所有染色质远程交互信息,还可以查询两两基因之间拥有的多层级交互基因网络,为水稻多基因之间共转录、共调控提供参考(图2)。
总之,该数据库全面展示了多维度的水稻表观基因组数据,涵盖了水稻不同品系不同组织间的表观基因组动态变化模式,为水稻功能基因组研究领域提供了解析水稻表观基因组和染色质远程互作信息的重要研究平台。
华中农业大学李国亮教授和李兴旺教授为通讯作者,博士生谢亮为文章第一作者。该研究得到国家重点研发计划,国家自然科学基金和华中农业大学自主科技创新基金的支持。
责任编辑:小邱同学
